O microbioma cutâneo: modelos e ensaios in vitro
Graça à sua experiência em engenharia celular e de tecidos, a QIMA Life Sciences desenvolveu modelos de estudo in vitro ou ex vivo que são adequados para a análise das interações entre microbiota e a pele:
- Cultura bacteriana isolada (Staphylococcus aureus, Staphylococcus epidermidis, Propionibacterium acnes)
- experimentos de infecção com vírus HSV-1 (vírus da Herpes simplex tipo I)
- Cocultura:
- bactérias em modelos de pele 2D (queratinócitos, fibroblastos, sebócitos etc.)
- bactérias em modelos de pele 3D (explantes, epiderme reconstruída etc.)
O resultado dos compostos ou das formulações sobre o desenvolvimento das bactérias e a influência das bactérias na resposta cutânea podem ser avaliados por estes modelos.
A QIMA Life Sciences oferece uma série de soluções para a avaliação de:
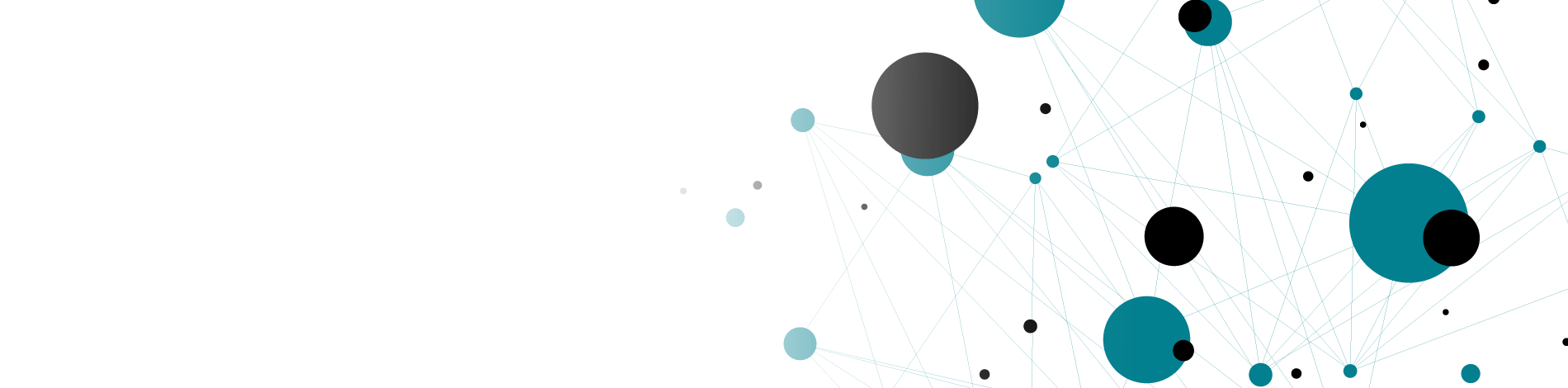
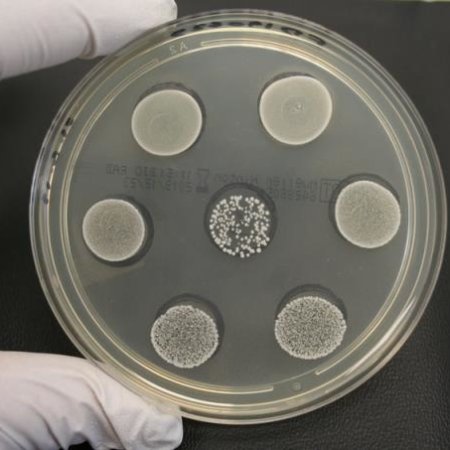
- Viabilidade bacteriana
- Quantificação da colônia bacteriana (CFU)
- Determinação da carga bacteriana [qPCR (Taqman)]
- Adesão bacteriana nos modelos 3D (explantes, epiderme reconstruída)
- Resposta biológica de modelos 2D ou 3D (diferenciação, inflamação e resposta imunológica)
Aqui se encontram alguns exemplos dentre todos os ensaios propostos pela QIMA Life Sciences sobre microbioma cutâneo:




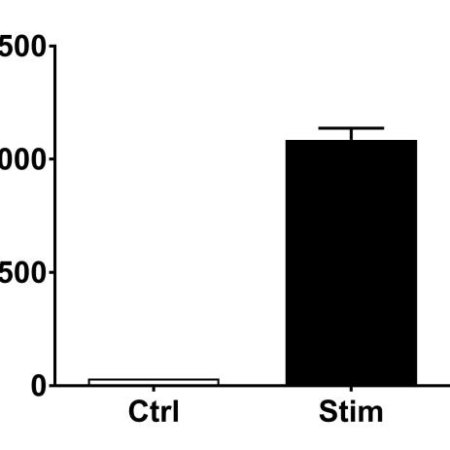
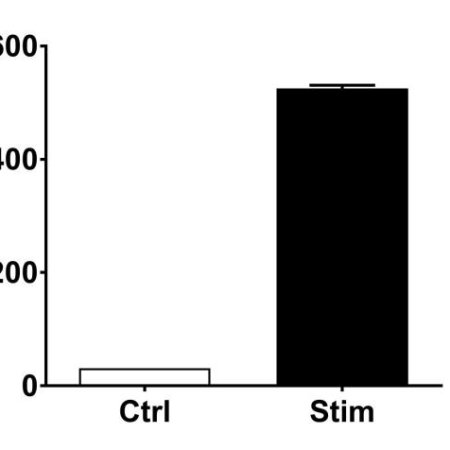


The Relationship Between Microbiology Testing and the Skin Microbiota
Cosmetics_cat_publi, Skin microbiomeMicrobiology testing is essential for substantiating microbiome-friendly and anti-microbial claims. Read on to find out how cosmetic microbiology testing is connected to the skin microbiome.
Microbiota vs. Microbiome: What’s the Difference?
Skin microbiomeWhat is the difference between microbiota vs. microbiome? Why are they important to human skin? What is the Skin Microbiota test? Learn more here.
Using Reconstructed Human Epidermis model in Skin Microbiome Research
Skin microbiome, Skin microbiomeSkin microbiome research is developing rapidly. Are you up to date? Find out why 3D skin models are an important development in skin microbiome research.
Profiling TLR agonist activity in keratinocytes and link to bacteria-induced innate immunity activation
Cosmetics_cat_publi, Microbiome cutané, Skin microbiomeIn this study, our goal was first to functionally profile and characterize the response of normal human keratinocytes (NHEK) to microbial patterns (TLR agonists) and IL-1α in parallel. To do so, the effects of multiple TLR agonists and IL-1α were evaluated on the gene expression profile, on signal transduction (NF-ƘB translocation) and also on IL-8 and IL-6 release. Secondly, we also tried to link these responses to those of pathogens of interest in skin microbiota (namely, C. acnes, S. aureus and P. aeruginosa), by evaluating the effect on IL-8 release by NHEK infected with living bacteria from these 3 strains…