Analyse du miRNome par microarray Affymetrix :
Bioalternatives propose différentes solutions pour l’analyse des miRNA. Une analyse du profil d’expression complet des miRNA (miRNome) à partir d’une puce à ADN/microarray permettra la mise en évidence de signatures moléculaires et l’identification de miRNA d’intérêts.
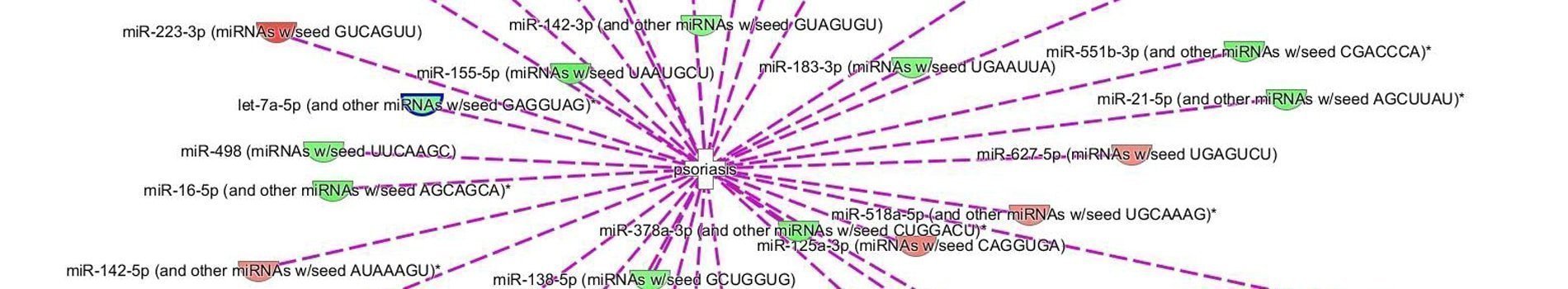
L’analyse du miRNome à partir de la puce miRNA_4.1 de Affymetrix va permettre d’identifier les miRNA significativement modulés à partir de calcul de fold change et de test statistique (t-test, réalisable en présence de réplicats), afin de mettre en évidence une signature moléculaire caractéristique de l’échantillon étudié (Figure 1). Une seconde étape d’analyse possible consiste à identifier les gènes cibles des miRNA significativement modulés à l’aide du logiciel IPA® de Qiagen (Figure 2).
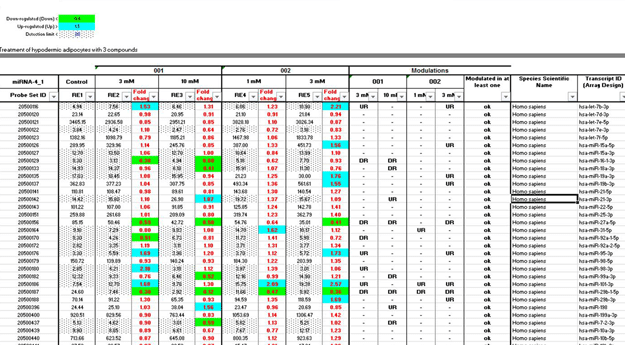
Figure 1 : Tableau d’analyse des résultats
La matrice de calcul de ce tableau permet de calculer les valeurs de fold change et de mettre ainsi en évidence les miRNA significativement modulés.
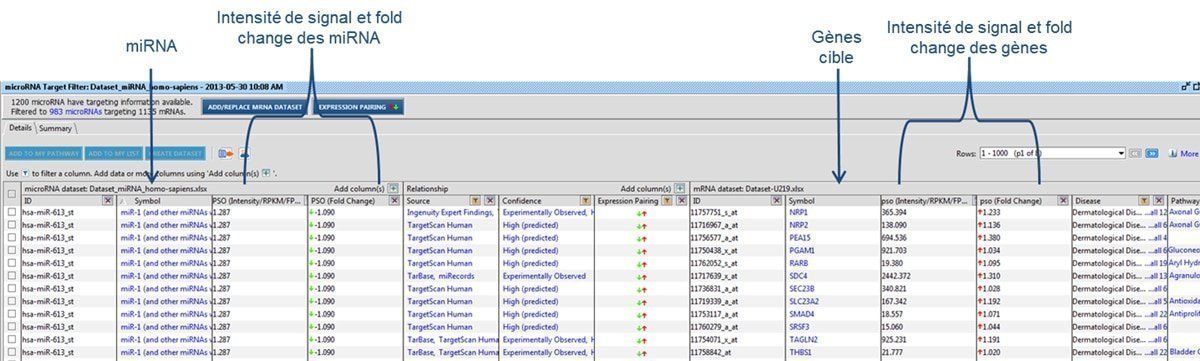
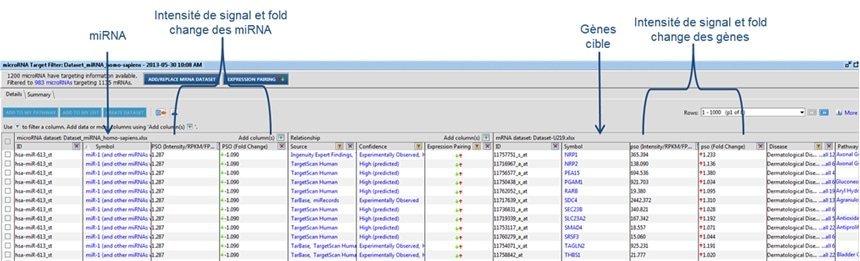
Figure 2 : Identification des gènes ciblés
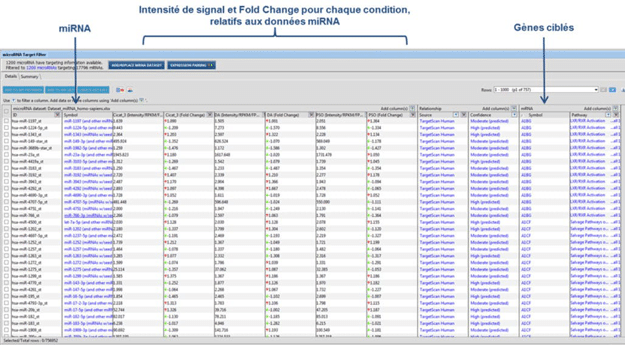
Dans le cadre d’études de mécanismes de régulation passant par les miRNA, l’analyse du miRNome peut être menée en parallèle d’une analyse du transcriptome réalisée à partir d’une puce U219 (puce humaine), avec les mêmes échantillons. L’hybridation d’un même échantillon sur ces deux types de puces va permettre d’identifier parmi les gènes significativement modulés, ceux étant potentiellement régulés par des miRNA. La correspondance entre les données issues des deux puces est réalisée à partir du logiciel IPA® de Qiagen (Figure 3).
Figure 3 : Correspondance entre les données issues des puces U219 et miRNA_4-1
Analyse de l’expression des miRNA par RT-qPCR :
Les expériences de RT-qPCR permettent d’analyser et de comparer le niveau d’expression de gènes d’intérêt dans des conditions données. Cette même technique peut être utilisée dans le cadre de l’analyse des miRNA.
Bioalternatives propose de réaliser l’extraction et l’analyse de l’expression de miRNA d’intérêt par RT-qPCR.
Applications :
- Validation de biomarqueurs
- Diagnostic et pronostic
- Recherche de gènes cibles
- Confirmation d’analyses par microarrays
Caractéristiques :
- Haute sensibilité
- Haute spécificité
- Rapidité
Principe :
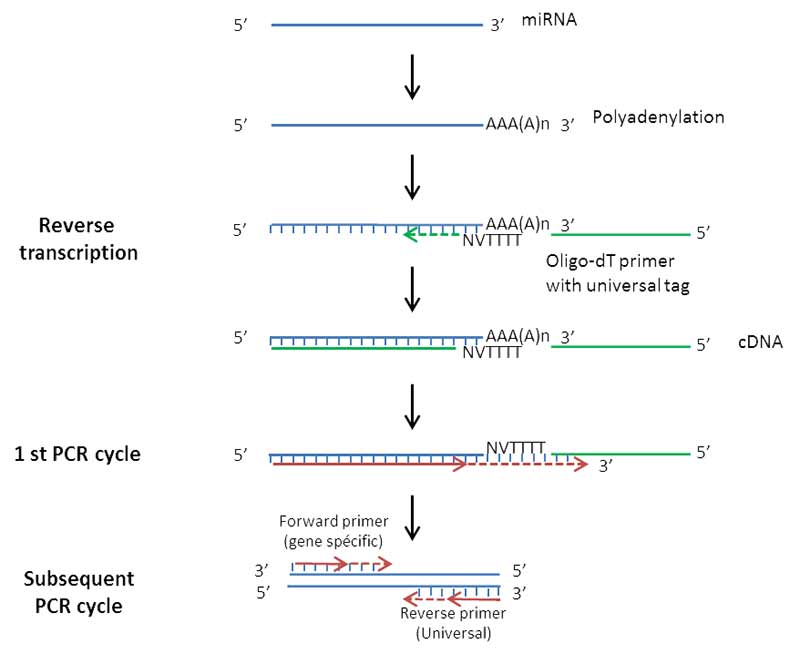
- Extraction des ARN totaux et purification des miRNA sur colonne
- Polyadénylation de miRNA
- Reverse transcription à l’aide d’un primer Oligo-dT
- Amplification par PCR à partir d’une amorce spécifique et d’une amorce universelle
- Mesure de l’incorporation des molécules fluorescentes
Figure 1: Principe d’analyse des miRNA par RT-qPCR
Analyse et interprétations des résultats :
L’analyse des résultats est basée sur une mesure en continue de l’incorporation de molécules fluorescentes (SYBR Green) au cours du processus d’amplification de la réaction de PCR. Ces mesures sont retranscrites en valeurs numériques (nombre de cycles) pour chaque gène. Ces valeurs sont ensuite transférées et traitées sous le logiciel Microsoft Excel afin de calculer le niveau d’expression relatif par rapport à une condition contrôle (Figure 2).
Pour aller plus loin, Bioalternatives possède les outils bio-informatiques et le savoir-faire nécessaires afin d’identifier les gènes cibles des miRNA d’intérêt.
Figure 2: Identification des gènes ciblés